

von Prof. Dr. Wilfried Brade (Hannover/Dummerstorf) und Prof. Dr. Ottmar Distl (Hannover)
Die Herausbildung des ruminalen Mikrobioms kann als eine
in jeder Generation neu stattfindende "Infektion" des Pansens
mit mikrobiellen Keimen interpretiert werden.
Der Pansen enthält ein sehr komplexes Ökosystem bestehend aus Mitgliedern aller drei Domänen des Lebens: Bacteria, Archaea und Eukarya (3, 20).
Die Vielzahl der Mikroorganismen im Pansen kann als ein Host-spezifisches Organ (ruminales Mikrobiom) aufgefasst werden, welches in den Wirt integriert ist (3).
Im Laufe des Lebens oder bei veränderter Fütterung/Nutzung des Wirtes kommt es zu strukturellen und funktionellen Veränderungen des ruminalen Mikrobioms.
Zu nennen sind hier deutliche Unterschiede im Mikrobiom eines juvenilen Rindes (praeruminales Kalb), einer kraftfutterreich ernährten Milchkuh oder eines raufutterreich ernährten Mastrindes.
Neueste Daten haben das Virom als weitere Komplexitätsebene diesem Konzept hinzugefügt (28). So können lytische Phagen1 die Bakterienanzahl und -struktur im Pansen beeinflussen.
In der Vergangenheit wurde die Struktur und Vielfalt der ruminalen Mikroorganismen vorrangig durch klassische mikrobiologische Techniken identifiziert (= In vitro-Kulturen). Mittels kulturabhängiger Methoden wird jedoch die tatsächliche mikrobielle Vielfalt im Pansen unterschätzt, da nur ein Bruchteil der vorhandenen Mikroorganismen kultivierbar ist (3, 5, 33).
Angeregt durch permanente Forderungen zur weiteren Verbesserung der Futtereffizienz von Rindern – bei gleichzeitig angestrebter direkter Einflussnahme auf eine möglichst reduzierte Methanbildung im Pansen – einschließlich rasanter Entwicklungen in der Molekulargenetik (Entwicklung genetische Marker) sowie in der Humanmedizin (= nachgewiesene Wechselwirkungen zwischen dem humanen Darmmikrobiom mit einer Vielzahl von speziellen Krankheiten wie Adipositas, Diabetes mellitus (Typ 2) oder Arteriosklerose – (17)), interessieren sich nun auch zunehmend die Tierwissenschaftler sowohl für die Interaktionen zwischen Genom x Ernährung als auch Genom x Genom (Wirt-Mikrobiom).
Jedes Individuum beherbergt dabei offensichtlich seine ureigensten Kommensalen2 (3, 26, 31). So ist bei Menschen die Zusammensetzung der Darmbakterien nicht völlig zufällig, denn die Host-spezifischen Bakterien können bestimmten Gemeinschaften zugeordnet werden, sogenannten Enterotypen. Diese wurden nach den jeweils dominanten Gattungen im Stuhlgang benannt: Enterotyp 1 (Bacteroides), Enterotyp 2 (Prevotella) und Enterotyp 3 (Ruminococcus).
Seither werden gewaltige Forschungsanstrengungen unternommen, das menschliche Mikrobiom zu kartieren. Internationale Mikrobiom-Projekte entstanden, so zum Beispiel das Human Microbiome Project (HMP) des US-amerikanischen National Institutes of Health (NIH), das 2008 gestartet wurde (13, 26). Während in der 1. Phase (bis 2012) die Mikroben vieler Körperbereiche charakterisiert wurden, werden in der aktuellen Phase deren biologische Eigenschaften untersucht und Kohortenstudien durchgeführt, um Zusammenhänge zwischen den Mikrobiota und Krankheiten herauszufinden.
Bei der Erforschung des Pansenmikrobioms der Wiederkäuer stehen wir demgegenüber erst am Anfang intensiver wissenschaftlicher Studien. Hier wurde in 2011 das "Hungate1000-Projekt"3 zur Etablierung der ruminalen mikrobiellen Genomik-Forschung initiiert. Diese globale Forschungsallianz vereint 27 Institute und Universitäten aus elf Ländern mit dem Ziel, einen Referenzsatz von mikrobiellen Pansensequenzen zu erzeugen, Strategien zur Effizienzverbesserung der Fermentation im Pansen zu erarbeiten und mögliche Minderungsstrategien bei der Umweltbelastung (zum Beispiel Minderung der Methanproduktion) aufzuzeigen (www.globalresearchalliance.org/).
Leider sind in diesem internationalen Projekt renommierte wissenschaftliche Einrichtungen aus dem deutschsprachigen Raum unterrepräsentiert.
Kultivierbare Mikroorganismen stellen nur einen winzigen Bruchteil der mikrobiologischen Gemeinschaft im Pansen und damit deren Vielfalt dar (3, 5, 24, 33).
Um Bakterien mittels einer kultivierunabhängigen Methode zu identifizieren, werden zunehmend phylogenetische Markermoleküle45 genutzt. Ein geeignetes Markermolekül wurde in der ribosomalen RNA gefunden (= 16S rRNA – (32)). Die Entwicklung weiterer Markersequenzen folgte (zum Beispiel die 18S rRNAs bei Eukaryoten).
Mittels der fast zeitgleich entwickelten PCR-Technik (polymerase chain reaction) war darüber hinaus eine Möglichkeit geschaffen, ausgesuchte Gene (oder Genfragmente) aus einem DNA-Gemisch in vitro zu vervielfältigen und einer genauen Untersuchung zuzuführen.
Weitere molekularbiologische Entwicklungen erleichtern die kulturunabhängige Identifikation von Prokaryoten zusätzlich (3, 5, 15, 25, 33).
Kulturunabhängige Analysen bakterieller rRNA-Gene bieten einen umfassenden Einblick in die Zusammensetzung gemischter mikrobieller Gemeinschaften in verschiedenen Habitaten (Böden, Biogasanlagen, Tiere und weitere).
Die Bestimmung der speziesspezifischen rRNA hat sich als verlässlicher phylogenetischer Marker erwiesen. Zwischenzeitlich wird die molekulargenetische Analyse komplexer rRNA-Sequenzen routinemäßig weltweit angewandt.
Öffentlich zugängliche Datenbanken mit umfangreichen Datensätzen dienen der schnellen Auswertung und phylogenetischen Zuordnung von rRNA-Sequenzen (zum Beispiel: www.arb-silva.de/news/view/2009/03/27/editorial/).
Die rRNA-Technik bietet also eine Möglichkeit der Identifikation und Klassifikation von Mikroorganismen, welche sonst nur erschwert oder gar nicht kultivierbar sind.
Jedoch hat auch diese Methode einige Einschränkungen (zum Beispiel, wenn es in der Aufbereitung nicht gelingt, Zellen adäquat zu lysieren). Auch vermögen die kulturunabhängige Methoden nicht zwischen lebenden und toten Mikroben zu unterscheiden.
Die Mikrobiologen haben deshalb – um die genomische Einzigartigkeit eines molekulargenetisch identifizierten, aber nicht genau taxonomisch zuordenbaren Organismus zu beschreiben – den Begriff "operative taxonomische Einheit (OTU)"6 oder auch "Phylotyp" eingeführt.
Der Pansen ist eine offene Fermentationskammer, deren Mikrobiom äußerst komplex ist. Die hier vorhandene Symbiose zwischen dem Wiederkäuer (Wirt) und seinen Mikroorganismen ist für beide Partner von Vorteil (3, 7, 24, 31).
Bakterien sind die quantitativ bedeutendsten Mitglieder der mikrobiellen Community.
(20) machen Angaben über den relativen Anteil verschiedener Domänen in Pansenproben bei Milchkühen und Mastochsen (bei unterschiedlicher Fütterung) sowie bei Ziegen oder Schafen (Abbildung 1). Die bakteriellen, eukaryotischen oder zu den Archaeen gehörigen rRNA-Anteile an der Gesamt-rRNA im Gastrointestinaltrakt schwanken zwischen den Tieren – innerhalb einer Art – erheblich. So variierte der Bacteria-Anteil zwischen 46,2 Prozent bis 98,2 Prozent, der Eukarya-Anteil zwischen 2,5 Prozent und 30,3 Prozent und der Archaea-Anteil zwischen 0,9 Prozent und 2,2 Prozent innerhalb der acht untersuchten Milchkühe (20). Ähnlich variierte der Anteil der verschiedenen Domänen bei den untersuchten Ziegen oder Schafen.
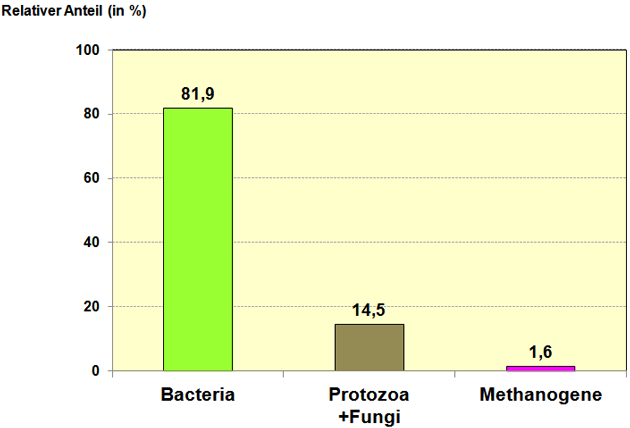
Abbildung 1: Relativer Anteil verschiedener Domänen am ruminalen Mikrobiom (in Anlehnung an (20)).
Quelle: Eigene Darstellung (hier verwendetes Datenmaterial: 50 Prozent Kraftfutteranteil an der Ration).
Funktionell lassen sich die ruminalen Mikroben entsprechend ihrer bevorzugten Energiequelle unterscheiden (7, 11, 31, 33). So bauen die cellulolytischen Bakterien zur Energieversorgung Cellulose ab, wachsen langsam und nutzen Ammoniak als Stickstoffquelle (Tabelle 1).Eine weitere, oft gewählte Einteilung der Ruminalbakterien erfolgt entsprechend ihres Vorhandenseins im Pansen (Abbildung 2):
Diese drei ruminalen "Habitats-Gruppen" interagieren im Pansen intensiv miteinander (10, 19, 21, 31, 33). Auch kommen sie in unterschiedlicher Häufigkeit vor. So weisen die Epithel-assoziierten Bakterien, die auch als epimurale Bakterien bekannt sind, in der Regel die geringste Dichte (im Vergleich zu den anderen beiden Populationstypen) aus.
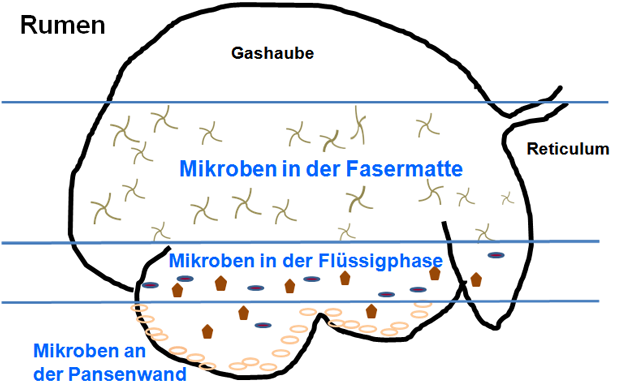
Abbildung 2: Mikrobenverteilung im Pansen.
Quelle: Eigene Grafik.
Besonders häufig zu findende Bakterienstämme im Pansen von Wiederkäuern sind Bacteroidetes8 und Firmicutes9 (14, 20, 25, 33).
Es gibt zwei Arten von Protozoen im Pansen; gegeißelte Protozoa (Urtierchen) oder bewimperte Protozoa. Die gegeißelten Protozoen überschreiten selten 103 Species pro Gramm Inhalt und ihr Beitrag zur Pansenfermentation ist eher unbedeutend (4, 24, 31, 33). Die bewimperten Protozoen bilden demgegenüber einen wesentlichen Teil der mikrobiellen Zellmasse und umfassen eine Vielzahl morphologischer Typen.
In manchen Wimpertierchen (Ciliaten) sind Hydrogenosomen vorhanden; spezielle Organellen (häufig anstelle von Mitochondrien), die ein Überleben unter anaeroben Bedingungen ermöglichen. Diese Protozoen-Species produzieren Wasserstoff (H2). Die Entfernung dieses Wasserstoffs erfolgt wiederum durch Methanogene (Archaeen). Daher führt die "künstliche" Beseitigung der Wimpertierchen (Defaunation) zu einer reduzierten Methanproduktion (3, 4, 6, 24).
Gut bekannt ist auch, dass Protozoen "Räuber" von Bakterien und anderen Mikroorganismen sind. Sie greifen somit regulierend in die Bakterienpopulationen ein. Eine Veränderung im Bestand oder der Zusammensetzung der Protozoen wirkt sich folglich auf die gesamte Mikrobengemeinschaft im Pansen aus (3, 6, 24).
Da Wimpertierchen (Ciliaten) regelmäßig Bakterien verdauen, könnten auch Teile dieser DNA, die von den Ciliaten aufgenommen wurde, in ihr Genom (in früheren evolutionären Entwicklungsphasen) integriert worden sein. Zum Beispiel besitzt die Xylanase10 produzierende Protozoen-Species Polyplastron multivesiculatum hier auffallend enge molekulargenetische Sequenzähnlichkeiten zu einer grampositiven Bakterien-Familie, die gleichfalls Xylanase produziert (6, 21, 24, 25).
| Tabelle 1: Ausgewählte Organismengruppen, gruppiert nach ruminalen Funktionstypen | |
|---|---|
| Gruppierung nach Funktionstyp | einige ausgewählte Vertreter (Arten) |
| Cellulose- und Hemicellulose-Abbauer (Funktionstyp: cellulolytisch) | Pansenbakterien: Ruminococcus albus, Butyrivibrio fibrisolvens (wichtige Gärendprodukte: Succinat, Acetat, Formiat, Butyrat, H2, CO2) |
| Stärke- und Zucker-Abbauer (Funktionstyp: amylolytisch) | Pansenbakterien: Selenomonas ruminantium, Bacteroides ruminicola (wichtige Gärendprodukte: Formiat, Acetat, Succinat, Laktat) |
| Laktat-Abbauer (Funktionstyp: lactolytisch) (Laktat → Propionat + Acetat) | Pansenbakterien: Selenomonas lactilytica, Veillonella parvula (Gärendprodukte: Propionat, Acetat, Butyrat, Succinat, CO2, H2) |
| Succinat-Abbauer (Funktionstyp: succinolytisch) (Succinat → Propionat + CO2) | Pansenbakterien: Schwartzia succinivorans, Veillonella parvula (Gärendprodukte: Propionat, CO2) |
| Pectin-Abbauer (Funktionstyp: pektinolytisch) | Pansenbakterien: Lachnospira multipara (Gärendprodukte: Acetat, Formiat, Lactat, H2, CO2) |
| Methanbildner* (CO2 + H2 → CH4); alternativ: Formiat → Methan | Methanogene aus der Domäne der Archaeen, zum Beispiel Methanobrevibacter ruminantium |
Anmerkungen: * Methan wird nicht in einer Gärung sondern durch eine anaerobe "Carbonatatmung" gebildet.
Die Pilze im Pansen sind oft durch einen speziellen zweistufigen Lebenszyklus gekennzeichnet. In der zoosporigen Stufe entwickeln sich aus (gegeißelten) Sporen – befestigt an Futterpartikeln – anaerobe Pilze, die sich dann in einem vegetativen Stadium weiterentwickeln. Ihre Myzel-Strukturen wachsen in die Futterpartikel ein und sind dort verantwortlich für die Erzeugung von zum Beispiel hydrolytischen Enzymen (Tabelle 1).
Aufgrund des zweistufigen Lebenszyklus und der Fähigkeit der Myzel-Strukturen in die Futterpartikel zu wachsen, ist es nur schwer möglich, die Pilzzellmasse im Pansen exakt zu quantifizieren. Einige indirekte Schätzungen lassen vermuten, dass Pilze bis zu zehn Prozent der mikrobiellen Massen ausmachen können (24, 31).
Das Interesse an dem Vorhandensein von Archaeen im Pansen hat in den vergangenen Jahren in der ruminalen Mikrobiomforschung – vor allem aufgrund der Fähigkeit einiger Arten Methan (CH4) zu produzieren – erheblich zugenommen (3, 24).
CH4 wird durch methanogene Archaeen produziert; eine vielfältige Gruppe obligat anaerober Archaea. Ihre CH4-Synthese kann als Endprodukt ihrer speziellen Atmung angesehen werden.
Die CH4-Bildung ist aus zweierlei Hinsicht problematisch: CH4 führt einerseits zu einem Energieverlust zwischen fünf bis zwölf Prozent der aufgenommenen Energie (3, 10). Und zum anderen wird das Treibhausgas CH4 von ihnen erzeugt, das wiederum der Wirt in die Umwelt freisetzt.
Der Pansen beinhaltet zusätzlich ein komplexes Virom, deren Viren systematisch die Zellen von Eukaryoten oder Prokaryoten befallen.
So wird der Pansen von einer großen Anzahl von Bakteriophagen11 bewohnt, die wahrscheinlich dazu beitragen, dass eine "Homöostase" der mikrobiellen Population erreicht wird. Schätzungen nennen hier bis zu 1011 Phagenpartikel pro Gramm Panseninhalt (28).
Darüber hinaus gibt es Hinweise auf die Übertragung von genetischem Material (horizontaler Gentransfer) zwischen verschiedenen Bakterien-Species einerseits und/oder zwischen Bakterien und beispielsweise bewimperten Protozoen andererseits (6, 24).
Im Laufe des Lebens sowie bei veränderter Fütterung kommt es zu kontinuierlichen Verschiebungen in der Zusammensetzung des ruminalen Mikrobioms, die wiederum auch durch das Genom des Wirtes und weiteren Einflussgrößen (zum Beispiel Jahresverlauf, Hitzestress) bestimmt wird.So gibt es Unterschiede zwischen dem praeruminalen Mikrobiom eines "Tränke"-Kalbes und dem Mikrobiom eines adulten Tieres (Abbildung 3).
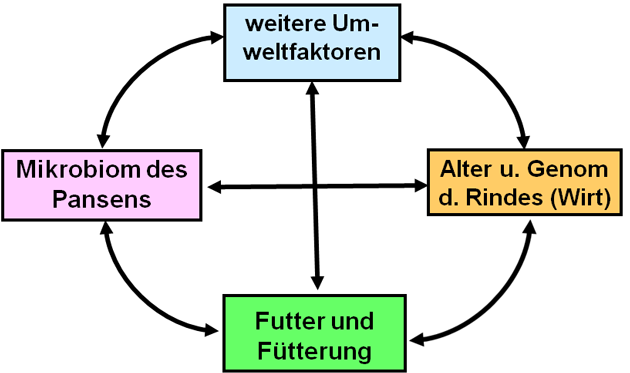
Abbildung 3: Wechselwirkungen zwischen verschiedenen Faktoren bei der Verdauung von Pflanzen oder Pflanzenteilen im Pansen eines Rindes.
Im "reifen" Pansen ausgewachsener Tiere ist die Besiedlung des Pansens nachhaltig von den eingesetzten Futtermitteln und damit der Nutzungsrichtung (zum Beispiel laktierende oder trockenstehende Milchkühe, Mastbullen) und dem besiedelten "Pansen"-Habitat einschließlich weiterer morphologisch-physiologischer Besonderheiten des Wirts (zum Beispiel Pansenvolumen) abhängig. Diese vielfältigen Einflüsse und Interaktionen sollen nachfolgend detaillierter beschrieben werden.
Bei Wiederkäuern ist der Gastrointestinaltrakt, einschließlich Vormagenkomplex, bei der Geburt steril. Allerdings wird er bereits in den ersten 24 Stunden nach der Geburt rasch mit Bakterien besiedelt. Die mikrobielle Dichte in der praeruminalen Pansenflüssigkeit erreicht schnell eine Konzentration von 109 Zellen/ml – beim Vorherrschen anaerober Bakterien – ab dem zweiten Tag nach der Geburt (11, 24). Gleichzeitig setzen anatomische Veränderungen, speziell in der Struktur der Pansenwand, ein. Der Haubenrinnenreflex12 bei Kälbern stellt sicher, dass die aufgenommene Milch auf kürzestem Weg direkt in den Labmagen transportiert wird, wo sie durch das Lab und die Salzsäure gerinnt. Während das Volumen des (noch) unterentwickelten Pansens nur etwa 29 Prozent des Gesamtmagenvolumens bei der Geburt eines Kalbes umfasst, steigt dieser Anteil – mit zunehmender Reife des Tieres – auf etwa 55 Prozent an (18).
Die praeruminalen Wandstrukturen – verbunden mit einer gleichzeitigen Verfügbarkeit von Nährstoffen – stellen offensichtlich vom zweiten Lebenstag an komfortable Nischen für die (Erst-)Besiedlung mit Mikroorganismen dar; neben proteolytischen Bakterien auch bereits für cellolytische Mikroben oder Methanogene.
Flagellaten (Protozoen) sind etwa ab der dritten Woche nach der Geburt zu beobachten (27).
Der Pansen-pH-Wert ist von entscheidender Bedeutung für die weitere Pansenentwicklung und Tiergesundheit. Pansenazidose ist eine mögliche Erkrankung bereits bei Kälbern; zum Beispiel beim Einsatz von ungeeigneten Milchaustauschern (zu schnelle Vergärung von Milch/ausgewählten Produkten in Milchaustauschern). Niedrige pH-Werte beeinflussen wiederum die Zusammensetzung der Mikrobiota.
(18) untersuchten die praeruminalen Mikrobiota von unterschiedlich alten männlichen Holstein-Kälbern (14 Tage (d) oder 42 Tage (d) nach der Geburt; gefüttert mit einem Standard-Milchaustauscher) und identifizierten das Vorkommen von 15 bakteriellen Phyla. Die mikrobiellen Communities wurden von den Stämmen Bacteroidetes, Firmicutes und Proteobakteria dominiert. Mit zunehmendem Alter der Kälber erhöhte sich der häufigste Stamm, Bacteroidetes, von 45,7 Prozent im Pansen der 14 Tage alten Kälbern auf 74,8 Prozent im Pansen der 42 Tage alten Kälbern (P < 0,1). Gleichzeitig nahm die relative Häufigkeit von Firmicutes, Proteobacteria sowie Fusobacteria ab (Abbildung 4). Bei einer Gegenüberstellung der Zusammensetzung der praeruminalen Mikrobiota im Vergleich zu der von 12 Monate (mo) alten Mastbullen konnten sie weitere Unterschiede belegen (18).
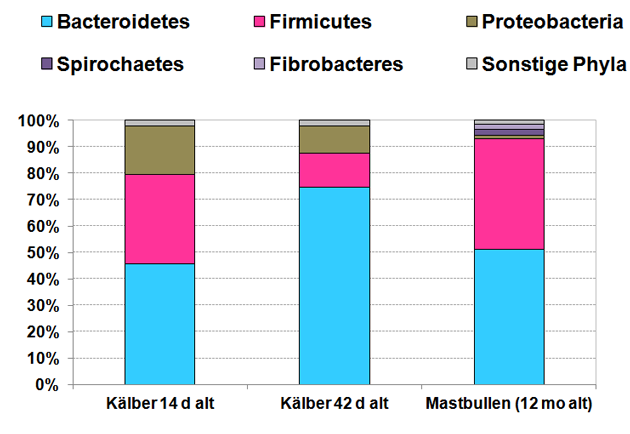
Abbildung 4: Relative Zusammensetzung der bakteriellen Mikrobiota von unterschiedlich alten Kälbern und einjährigen Mastbullen auf Stammebene (Phyla).
Quelle: (18) – eigene Darstellung.
Die vier am häufigsten vorkommenden Stämme im reifen Pansen der Bullenkälber (bei einer Heu-basierten Fütterung) waren Bacteroidetes (52,0 Prozent), Firmicutes (42,7 Prozent), Spirochäten (2,3 Prozent) und Fibrobacteres (1,9 Prozent) (Abbildung 4).
Eine Bewertung der Mikrobiota auf Genera-Ebene ergab das Vorhandensein von insgesamt 170 Bakteriengattungen bei den untersuchten praeruminalen Rindern; jedoch nur 45 Gattungen wurden in allen Proben gefunden. Diese Genera können als der Kern (Core) der praeruminalen bakteriellen Community angesehen werden.Auch auf Gattungsebene sind wiederum signifikante Häufigkeitsunterschiede in Abhängigkeit vom Alter der Rinder zu beobachten (Tabelle 2).
| Tabelle 2: Einige ausgewählte Gattungen mit signifikanten Häufigkeitsunterschieden in den Mikrobiota unterschiedlich alter Kälber | ||
|---|---|---|
| Genus | Relative Häufigkeit in Prozent | |
| 14 Tage alte Kälber | 42 Tage alte Kälber | |
| Bacteriodes | 18,080 ± 14,758 | 71,376 ± 15,151 |
| Fusobacterium | 0,993 ± 0,397 | 0,023 ± 0,019 |
| Ruminococcus | 0,265 ± 0,102 | 0,020 ± 0,015 |
| Luteimonas | 0,006 ± 0,003 | 0,000 ± 0,000 |
| Mogibacterium | 0,055 ± 0,027 | 0,000 ± 0,000 |
Quelle: (18, stark gekürzt).
Im Pansenmikrobiom der 14 Tage alten Kälber war die Gattung Prevotella mit 33,1 Prozent klar dominierend; gefolgt von Bacteroides (18,1 Prozent), Oscillibacter (12,9 Prozent), Paraprevotella (7,1 Prozent) und andere. Es gab 13 Gattungen mit einer relativen Häufigkeit von mehr als einem Prozent in der bakteriellen Gemeinschaft der 14 Tage alten Kälber. Einige Genera (zum Beispiel Arcanobacterium, Flavobacterium und Mogibacterium) waren bei den jungen Kälbern vorhanden; jedoch nicht mehr bei den älteren Tieren (Tabelle 2).
Insgesamt bestätigt die Untersuchung von (18) die enorme Heterogenität der Pansenmikroflora auf Genera-Ebene bereits bei sehr jungen Kälbern. Gleichzeitig konnte auf Gattungsebene ein bemerkenswerter Anstieg der Bacteroides von 18,1 Prozent im Pansen von 14 Tage alten Kälbern auf 71,4 Prozent bei 42 Tage alten Kälbern beobachtet werden (Tabelle 2).
Zwischenfazit: Das Pansenmikrobiom der jüngeren Kälber (14 Tage) zeigt eine heterogenere mikrobielle Zusammensetzung und beherbergt – zumindest vorübergehend – eine größere Zahl von Bakterien-Gattungen als ältere Kälber (42 Tage); trotz gleicher Diät (ad libitum Fütterung des gleichen Milchaustauschers). Festzuhalten bleibt zusätzlich, dass bemerkenswert stabile Stoffwechselpotentiale – sowohl bei sehr jungen (14 Tage) als auch älteren Kälbern (42 Tage) – nachzuweisen sind (18).
Auch (34) zeigen, dass die ruminalen Bakteriengemeinschaften bei Kälbern, Milchkühen oder Mastbullen deutlich verschieden waren; bei einer beeindruckenden mikrobiellen Vielfalt und Komplexität in allen drei Tiergruppen (Tabelle 3).
| Tabelle 3: Mittlere Anzahl nachgewiesener bakterieller Taxa oder OTU’s im Pansen in Abhängigkeit vom Alter oder der Nutzungsrichtung/Rasse; (in Klammern: Standardfehler) | |||||
|---|---|---|---|---|---|
| Tiergruppe/ Untersuchungsmaterial | Taxa | OTUs* | |||
| Stamm (Phylum) | Klasse (Clasis) | Familie (Familia) | Gattung (Genus) | ||
| Kälber (HF-Bullenkälber, etwa 42 Tage alt) | 12,3 | 20,0 | 49,0 | 97,0 | 133,3 |
| "reifer" Pansen bei laktierenden Milchkühen (HF-Kühe) | 17,3 | 22,8 | 41,0 | 73,8 | 537,5 |
| Mastbullen (rund 270 kg) | 15,6 | 22,3 | 39,4 | 76,5 | 499,6 |
| gesamt | 21 | 31 | 93 | 219 | 1.079 |
Quelle: (34, stark gekürzt).
Anmerkungen: * operative taxonomische Einheiten.
Sie konnten insgesamt 21 Stämme, 31 Klassen, 93 Familien und 219 verschiedene Gattungen nachweisen (Tabelle 3). Insgesamt belegt diese Studie: Während die Anzahl der Stämme im sich entwickelnden Pansen im Mittel signifikant kleiner als im reifen Pansen war, konnte auf Gattungsebene demgegenüber bei den praeruminalen Tieren (Kälbern) im Mittel eine größere Anzahl vorhandener Gattungen identifiziert werden (Tabelle 3).
Bei Wiederkäuern mit einem reifen Pansen gelangen die Futtermittel – nach flüchtigem Kauen – mit dem Abschlucken in den Hauben-Pansen-Raum (16). Dort ist – wie bereits beschrieben – eine Schichtung (Gasschicht, Faserschicht Flüssigkeitsschicht) gegeben (Abbildung 2).
Das Wiederkäuen ist ein angeborener Vorgang. Die physikalische Struktur des Vormageninhaltes wird als wichtiger Reiz für die Auslösung des Wiederkäuens angesehen (Abbildung 5).
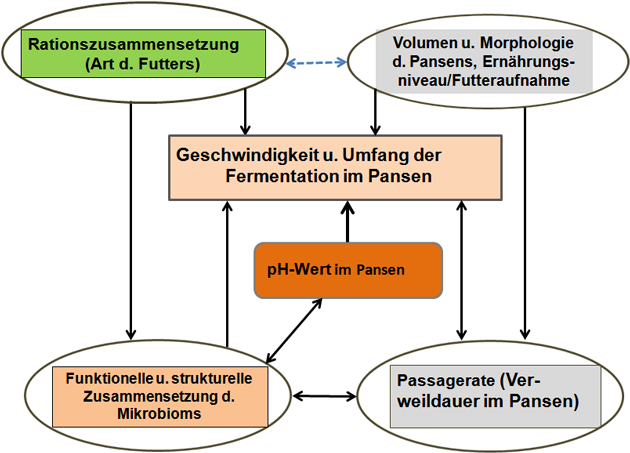
Abbildung 5: Beziehungen zwischen Rationszusammensetzung, Mikrobiom, Pansenmorphologie und Ernährungsniveau.
Quelle: Eigene, schematische Darstellung in Anlehnung an (16).
Die mikrobiellen Umsetzungen des Futters im Pansen verändern die Zusammensetzung der vom Wirtstier aufgenommenen Nahrung grundlegend (16, 31). Kann eine Spezies im Pansen ihr Stoffwechselprodukt nicht selbst weiterverwerten, wird dieses in der Regel sofort von einer anderen Arten der ruminalen mikrobiellen Gemeinschaft weiter genutzt. Der Umfang des mikrobiellen Abbaus im Pansen ist von der Abbaugeschwindigkeit sowie der Verweildauer des Futtermittels im Pansen abhängig (Abbildung 5). Während die Verweildauer durch die Höhe der Futteraufnahme und das tierseitig vorhandene Pansenvolumen modifiziert wird, hängt die Geschwindigkeit des Abbaus beispielsweise der Kohlenhydrate in den Vormägen weitgehend von der Art einschließlich der technischen Aufbereitung (mechanische Zerkleinerung) der verabreichten Futtermittel (zellulosereiche oder stärkereiche Fütterung) ab (Abbildung 6).
Zusätzlich verändern sich sowohl Struktur als auch Transportfunktionen des Pansenepithels von Wiederkäuern in Abhängigkeit von der Fütterung. Bei vermehrter Aufnahme von Kraftfutter, die erhebliche Veränderungen der Pansenflüssigkeit nach sich zieht (erhöhte Konzentration von kurzkettigen Fettsäuren, pH-Absenkung und anderes), nimmt bekanntermaßen die Anzahl und Größe der Zotten zu, die Verhornung wird stärker und Transportleistungen für Ionen werden erhöht (30). Der Ablauf und die Regulation dieser Adaptationsvorgänge werden allerdings bislang nur unzureichend verstanden.
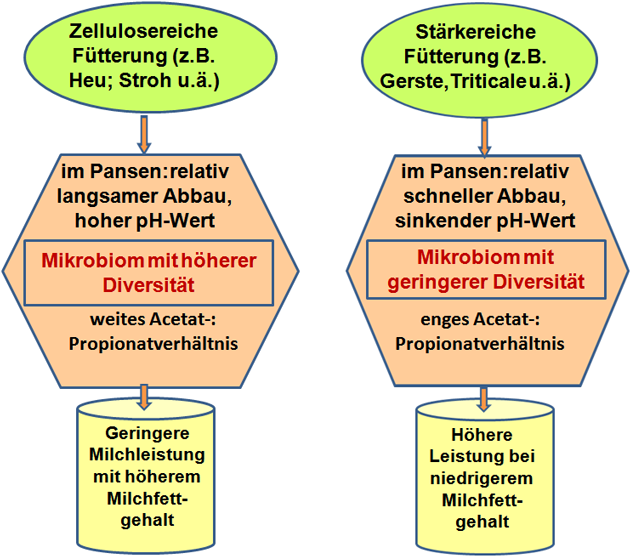
Abbildung 6: Schematische Darstellung möglicher Veränderungen im Pansen in Abhängigkeit von der Rationsgestaltung.
Von besonderer Bedeutung für den Umfang der Umsetzungen in den Vormägen ist, vor allem bei Milchkühen, die Verweildauer des aufgenommenen Futters im Pansen, das heißt die Passagerate durch die Vormägen (16, 31). Generell reduziert sich mit steigender Futteraufnahme die Aufenthaltsdauer des Futters im Pansen, das heißt die Passagerate nimmt zu (Abbildung 6).
| Tabelle 4: Fütterungsniveau und Fermentationsprodukte von Milchkühen (vier Fütterungsvarianten) | ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Kenngröße | Fütterungsvariante | Probenahme | Signifikanz (P-Werte) | |||||||
| proteinreich1) | proteinarm1) | 0 h* | 2,5 h | 5 h | Protein- stufe | Energie- stufe | Zeit | |||
| FIB | STA | FIB | STA | |||||||
| Aufnahme (kg/d): | ||||||||||
| Organische Masse (OM) | 19,0 | 18,7 | 19,0 | 18,8 | 0,92 | 0,60 | ||||
| N | 0,44 | 0,43 | 0,34 | 0,33 | < 0,001 | 0,28 | ||||
| NDF (neutral detegent fiber) | 9,62 | 7,20 | 10,3 | 7,24 | 0,12 | < 0,001 | ||||
| Pansenfermentation: | ||||||||||
| Ammoniak (mmol/l) | 9,54 | 6,57 | 6,62 | 1,54 | 4,46b | 11,1a | 2,66b | < 0,001 | < 0,001 | < 0,001 |
| pH-Wert | 6,56 | 6,46 | 6,63 | 6,44 | 7,04a | 6,38b | 6,15b | 0,54 | < 0,001 | < 0,001 |
| Gesamt-VFA (mmol/l) | 107 | 102 | 102 | 101 | 75b | 113a | 120a | 0,20 | 0,28 | < 0,001 |
| Acetat (%) | 67,6 | 65,5 | 67,3 | 65,6 | 70,9a | 63,5b | 65,2b | 0,96 | 0,027 | < 0,001 |
| Propionat (%) | 18,0 | 17,4 | 18,5 | 17,0 | 15,6c | 20,0a | 17,5b | 0,93 | 0,008 | < 0,001 |
| Relative Häufigkeit verschiedener bakterieller Species (stark gekürzt): | ||||||||||
| R. flavefaciens | 3,01b | 4,13a | 1,89c | 4,61a | 0,06 | < 0,001 | ||||
| F. succinogenes | 11,6b | 13,5a | 10,5bc | 9,26c | < 0,001 | 0,43 | ||||
| B. fibrisolvens | 14,0 | 15,7 | 10,9 | 10,6 | < 0,001 | 0,93 | ||||
| S. ruminantium | 0,20 | 0,36 | 0,26 | 0,29 | 0,92 | 0,035 | ||||
| DNA-Konzentration weiterer Domänen: | ||||||||||
| Protozoa (mg/g T) | 0,71 | 0,47 | 0,54 | 0,44 | 0,021 | 0,005 | ||||
| anaerobe Pilze (µg/g T) | 1,70a | 1,40b | 1,58ab | 0,66c | < 0,001 | < 0,001 | ||||
| Methanogene (107 Kopien/g T) | 4,87 | 3,94 | 3,60 | 2,80 | < 0,001 | < 0,001 | ||||
Quelle: (2, stark gekürzt).
Anmerkungen: FIB = ballaststoffreiche Fütterung; STA = stärkereiche Fütterung; VFA = flüchtige Fettsäuren, 1) 110 % vs 80 % des Bedarfs; * 0 h = Probenahme vor der Fütterung, 2,5 h = Probenahme 2,5 Stunden nach Fütterung; T = Trockenmasse.
Bestätigt werden die dargestellten Zusammenhänge durch zahlreiche Fütterungsversuche. So zeigten beispielsweise (2), dass bei Verabreichung einer ballaststoffreichen Diät die Kühe einen höheren pH-Wert im Pansen, eine höhere Ammoniak-Konzentration und einen höheren Anteil an Acetat und Propionat gegenüber einer ballaststoffarmen Ration hatten (Tabelle 4).
Da der Panseninhalt der laktierenden Kühe (rund 70 Tage nach der Abkalbung mit einer mittleren Leistung von über 30 Kilogramm Milch/Tag) vor der Fütterung (0 h), 2,5 und fünf Stunden nach der Morgenfütterung mehrtätig analysiert wurde (Tabelle 4), konnten weitere interessante Ergebnisse belegt werden: ein Ammoniak-Peak ist etwa 2,5 Stunden nach der Fütterung vorhanden, der erwartungsgemäß bei proteinreicher Ernährung höher ausfällt. Die ruminale Konzentration an Gesamt-VFA13 oder der Anteil an Butyrat vergrößert sich nach der Fütterung und bleibt dann über die nächsten fünf Stunden konstant.
Zusätzlich prüften (2) die Wirkung einer Stickstoff-Unterernährung (110 Prozent versus 80 Prozent zum Bedarf) bei differenzierter Art der Kohlenhydratzufuhr [ballaststoffreich (FIB) versus stärkereich (STA)] auf das ruminale Ökosystem (Tabelle 4).
Eine ballaststoffreiche Fütterung erhöht die ruminale Bakterienvielfalt einschließlich die relativen Häufigkeit der Protozoen (+ 38 Prozent), anaeroben Pilze (+ 59 Prozent) und Methanogene (+ 27 Prozent); summa summarum diejenige von cellulolytischen Mikroorganismen.
Ausgewählte Mikroorganismen im Pansen reagierten gleichzeitig empfindlich auf einen Mangel an Stickstoff (Tabelle 4).
Das Gesamtergebnis bestätigt, dass das ruminale mikrobielle Ökosystem direkt mit dem Pansenfermentationsmuster gekoppelt ist.
Der Zeitraum rund drei Wochen vor dem Kalben bis etwa drei Wochen nach dem Abkalben, die sogenannte Transitphase, ist entscheidend für die Gesundheit und Leistungsfähigkeit einer Milchkuh. Während dieser Phase kommt es zu enormen Veränderungen im Stoffwechsel und Energiebedarf (einschließlich einer negativen Energiebilanz); verbunden mit einer veränderten Fütterung. Die mikrobielle Gemeinschaft reagiert dynamisch auf diese Veränderungen, das heißt im ruminalen Mikrobiom kommt es zu raschen strukturellen und funktionellen Modifikationen während dieses Übergangs von der nicht-laktierenden zur laktierenden Periode. Gleichzeitig verändert sich das Fütterungsregime (mit einem schnell zunehmendem Konzentrateinsatz).
(25) untersuchten den Panseninhalt von erstlaktierenden und multipaaren Kühen während der Transit-Periode (experimentell gewonnen mit der Schlundsonden-Methode). Sie konnten 17 verschiedene Bakterienstämme nachweisen.
Die am häufigsten vorkommenden Stämme waren Bacteroidetes und Firmicutes (Tabelle 5).
| Tabelle 5: Anteil verschiedener Bakterienstämme bei erstlaktierenden Kühen (Transit-Periode) | ||||
|---|---|---|---|---|
| Stamm (Phylum) | erstlaktierende Kühe | |||
| drei Wochen vor der Kalbung | ein bis drei Tage nach der Kalbung | vierte Laktationswoche | achte Laktationswoche | |
| Bacteriodetes | 80,8 | 91,2 | 86,5 | 87,9 |
| Fibrobacteres | 1,44 | 0,30 | 1,34 | 1,36 |
| Firmicutes | 12,61 | 5,94 | 8,60 | 6,35 |
| Proteobacteria | 0,99 | 0,44 | 0,40 | 1,59 |
| Tenericutes | 1,54 | 0,77 | 1,04 | 0,62 |
| Cyanobacteria | 0,66 | 0,06 | 0,47 | 0,30 |
| Spirochaetes | 0,12 | 0,07 | 0,12 | 0,19 |
Quelle: (25, stark gekürzt).
Das Verhältnis von Bacteroidetes zu Firmicutes änderte sich signifikant während der Transit-Periode (Zunahme des Anteils von Bacteroidetes und gleichzeitige Abnahme von Firmicutes (P < 0,05)).
Die dominierenden Gattung im Bacteroidetes-Stamm war Prevotella, dessen Anteil mit zunehmender Laktationsperiode deutlich zunahm; während der Anteil der Gattung Paludibacter (und mehrere andere) abnahm (25).
Wie bereits erwähnt, kann eine Einteilung der Bakterien des Pansens an Hand ihrer Lokalisation vorgenommen werden.
Die meisten bisher durchgeführten Untersuchungen konzentrierten sich auf die an den festen Partikeln anhaftenden oder in der Pansenflüssigkeit vorkommenden Bakterien. Gemeinsam ist allen, dass sie sensibel auf Veränderungen in der Fütterung, im Gesundheitszustand des Wirts oder auf eingesetzte Antibiotika reagieren (19, 24).
(35) untersuchte die Biodiversität der Bakteriengemeinschaften im Pansen von Kühen, die bevorzugt Luzerne oder alternativ Triticale (Getreide-Diät) erhielten, in drei getrennten Fraktionen:
Sie konnten 613 operative taxonomische Einheiten (OTUs) identifizieren, die 32 Gattungen, 19 Familien und neun Phyla (Stämme) der Domäne Bakteria zugeordnet werden konnten. Die bakterielle Diversität war erwartungsgemäß bei der Luzerne- im Vergleich zur Triticale-Fütterung vielfältiger und gleichzeitig innerhalb der drei Fraktionen im Pansen unterschiedlich (35).
(19) untersuchten die bakteriellen Gemeinschaften sowohl im Panseninhalt (RC) als auch am Pansengewebe (RT) von Kreuzungs-Mastrindern (n = 22).
| Tab. 6: Mittlere Anzahl beobachteter Bakterienkopien insgesamt oder der Gattung Prevotella | |||
|---|---|---|---|
| Kenngröße | Panseninhalt | Pansengewebe | Signifikanz (P-Wert) |
| Gesamtanzahl an Bakterienkopien (Anzahl Kopien/Gramm) | 5,87 x 1010 | 8,86 x 108 | < 0,0001 |
| Gattung Prevotella (Anzahl Kopien/Gramm | 4,04 x 1010 | 1,99 x 108 | < 0,0001 |
| Anteil der Gattung Prevotella ( in Prozent) | 60,12 | 21,32 | < 0,0001 |
Quelle: (19).
Die Gesamt 16S rRNA-Gen-Kopienzahl lag zwischen 6,18 x 108 bis 2,04 x 1011 Kopien pro Gramm Panseninhalt und zwischen 5,63 x 107 und 6,58 x 109 Kopien pro Gramm Pansengewebe (Tabelle 6).
Für das Genus Prevotella variierte die 16S rRNA-Gen-Kopienzahl unter den 22 Tieren von 4,60 x 108 bis 9,83 x 1010 Kopien pro Gamm Panseninhalt und zwischen 8,57 x 106 bis 1,40 x 109 Kopien pro Gramm Pansengewebe (Tabelle 6).
Die erhebliche Variation in der mikrobiellen Vielfalt oder Dichte zwischen den Individuen – sowohl in den bakteriellen RC- als auch RT-Gemeinden – bleibt festzuhalten (3, 19, 24, 33).
Detaillierte Untersuchungen der relativen Häufigkeit bestimmter Bakteriengruppen mit einem bestimmten Host-Phänotyp dürften helfen, die interindividuelle Variation zu erklären und mögliche Assoziationen zwischen der Höhe ausgewählter Nutzleistungen von Tieren zum vorhandenen Pansenmikrobiom aufzudecken.
Das generelle Interesse an möglichen Wechselwirkungen zwischen Wirt und seinem Mikrobiom erfuhr eine deutliche Zunahme als man eine Kopplung zwischen verschiedenen Mikrobiota im Darm und weiteren physiologischen Modifikationen bezüglich der Nahrungs-Energieverwertung sowohl bei Mäusen als auch Menschen nachweisen konnte (17).
Diskutiert wird aktuell auch die mögliche Bedeutung der frühen Darmflora bei Kindern und das Entstehen von ausgewählten Autoimmunkrankheiten oder metabolischen und neurologischen Erkrankungen des Menschen oder die Frage, wie die Darmflora das menschliche Immunsystem zu beeinflussen vermag (1, 17).
Bei Wiederkäuern liegen gleichfalls erste wissenschaftliche Hinweise zur Wechselwirkung des ruminalen Mikrobioms und dem Host-Genom vor.
(12) berichten, dass wachsende Rinder mit einer unterschiedlichen Futtereffizienz (differenzierte Restfutteraufnahme, RFI14) auch eine unterschiedliche Konzentration an flüchtigen Fettsäuren (VFA) im Pansen aufweisen.
Die bakteriellen DNA-Profile der 18 analysierten Pansensaft-Proben belegen zusätzlich, dass jedes Tier eine charakteristische Pansenbakteriengemeinschaft besitzt (12). Gleichzeitig bestätigten Ähnlichkeitsanalysen, dass die bakterielle Community im Pansen der effizienteren RFI-Ochsen einander ähnlicher war als die im Pansen von ineffizienteren RFI-Tieren.
(14) untersuchten laktierende Israelische Holsteins im Hinblick auf mögliche Interaktionen zwischen ihren Milchleistungen und ihren tierindividuellen Pansenmikrobiota (n = 15). Alle Kühe erhielten dieselbe Diät (bestehend aus 30 Prozent Ballaststoffen und 70 Prozent Konzentrat) ad libitum gefüttert. Den Kühen wurde Pansenflüssigkeit entnommen und weitere physiologische Parameter aufgezeichnet.
Insgesamt wurden 151 bakterielle Gattungen in den israelischen Pansenproben nachgewiesen. Interessant ist, dass die genannten Autoren eine positive Beziehung zwischen der täglichen Milchfettleistung (kg/d) und dem ruminalen Firmicutes : Bacteroidetes-Verhältnis fanden.
Auch wenn die Resultate von (14) eine bemerkenswerte Ähnlichkeit mit denen in anderen Säugerwirtssystemen (Interaktionen des Darm-Mikrobioms mit dem Wirtsphänotyp) erkennen lassen (siehe oben), bedarf es einer umfassenden weiteren Verifikation dieser ersten Ergebnisse.
Die Analyse fäkaler Metagenome von Menschen hat zwischenzeitlich in der Humanmedizin breiten Eingang gefunden. Auch besteht zwischenzeitlich im humanen Bereich die Möglichkeit (gegen Kostenbeteiligung) aus einer individuellen Stuhlprobe das mikrobielle Profil der Darmbakterien zu sequenzieren und so "sein individuelles Darmmikrobiom" analysieren zu lassen (Portal "my.microbes" des Europäischen Labors für Molekularbiologie (EMBL) Heidelberg).
Die mikrobielle Besiedlung in Kotproben repräsentiert das individuell vorhandene Mikrobiom am Ende des Gastrointestinaltraktes (Rectum).
Rektal gewonnene Kotproben von Rindern sind zwischenzeitlich wiederholt genutzt worden, um die tierindividuelle mikrobielle Besiedlung oder mögliche Effekte differenzierter Rationen auf das Mikrobiom (vergleichsweise einfach) zu erfassen (8, 9, 15, 29).
So untersuchten (15) die fäkale Mikrobiota unterschiedlich gefütterter Mastrinder (Tabelle 7).
Tabelle 7: Relativer Anteil dominanter Taxa in Faeces (Kotproben) von Mastrindern (verschiedene Fütterungsgruppen) | |||
|---|---|---|---|
| Taxa* | Relativer Anteil in den Fütterungsgruppen in Prozent | ||
| Getreideanteil: moderat | Getreideanteil: hoch | Grundfutteranteil: hoch | |
| Firmicutes | 50,31c | 76,91a | 70,15b |
| Oscillibacter | 4,57b | 8,10a | 0,65c |
| Turicibacter | 1,26c | 8,40a | 4,37b |
| Roseburia | 3,94a | 4,21a | 0,38b |
| Clostridium | 1,13b | 2,92a | 1,29b |
| Ruminococcus | 0,11b | 0,32a | 0,39a |
| Bacteroidetes | 37,39a | 12,82b | 1,83c |
| Prevotella | 14,39a | 2,15b | 0,09c |
| Bacteroides | 0,97a | 0,77a | 0,06b |
| Actinobacteria | 0,14c | 1,77b | 2,93a |
Quelle: (15, stark gekürzt).Anmerkungen: * Phylum jeweils fett gedruckt.
Die dominierenden Phyla waren in allen Kotproben wiederum Firmicutes und Bacteroidetes. Firmicutes war vor allem in der sehr kraftfutterreichen Fütterungsgruppe dominierend (Abbildung 7).
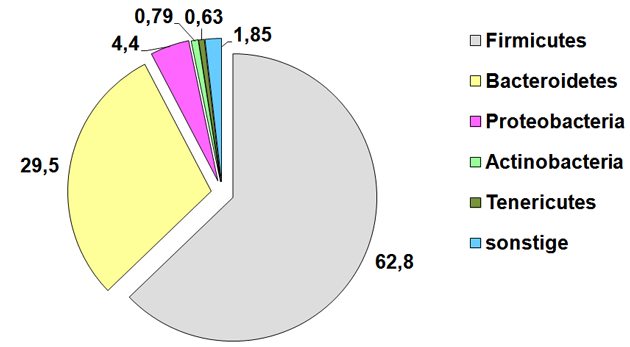
Abbildung 7: Prozentualer Anteil verschiedener Bakterienstämme in Kotproben von Mastrindern.
Quelle: (8) – eigene Darstellung.
Innerhalb des Phylums Firmicutes waren die Gattungen Oscillibacter, Turicibacter und Roseburia – mit signifikanten Unterschieden zwischen einzelnen Fütterungsgruppen – besonders häufig zu finden.
Insgesamt belegen die Untersuchungsergebnisse von (14), in Übereinstimmung mit (9), dass die fäkale Mikrobengesellschaft signifikante strukturelle Häufigkeitsunterschiede zwischen verschiedenen Fütterungsgruppen zu manifestieren vermag.
Durch die Entwicklung neuer molekularbiologischer Hochdurchsatzmethoden (zum Beispiel Next-Generation-Sequenzing) in Verbindung mit der Entwicklung phylogenetischer Marker sind nun erstmalig auch kulturunabhängige Einblicke in komplexe Mikrobiota möglich.
Aus der Humanmedizin ist bekannt, dass die Anzahl der Gene der an der Mensch-Mikroben-Symbiose beteiligten Mikroorganismen die des menschlichen Genoms um den Faktor 100 übersteigt (1, 13, 26). Für Wiederkäuer stehen derartige Bewertungen noch aus.
Seit über 2,6 Milliarden Jahren existieren Bakterien auf der Erde. Sie reagieren schnell auf Umweltänderungen. Bakterien sind "begnadete Chemiker" und deshalb für uns Menschen oder unsere Haustiere überlebenswichtig (22).
Und das besondere an ihnen: Bakterien bilden Netzwerke und kommunizieren untereinander.
Auch die ersten Nachweise über (bisher) ungeahnte Wechselwirkungen zwischen Wirt und Genom eröffneten Einblicke, die völlig neu sind.
Interessant ist, dass es nun auch erste Hinweise gibt, dass zwischen der täglichen Milchfettleistung (kg/d) bei hoch leistenden Milchkühen und dem ruminalen Firmicutes : Bacteoidetes-Verhältnis eine positive Assoziation gibt (Abschnitt 5).
Das Verhältnis von Bacteroidetes zu Firmicutes änderte sich auch signifikant während der Transit-Periode von Milchkühen (25).
Wir sind erst am Anfang, diese vielfältigen Wechselwirkungen zwischen Fütterung, ruminalen Mikrobiom und Wirt überhaupt zu erfassen.
Bakterien sind offensichtlich in ihrer Mehrzahl Freunde und nur ein kleiner Teil von ihnen vermeintliche Feinde (22).
Wiederkäuer sind bezüglich der Verdauung des Futters von ihren Mikrobiota abhängig.
Es ist daher verlockend, bereits eine Verbindung zwischen der Zusammensetzung und Menge verschiedener Pansenbakterien und physiologischen Parametern des Wirts zu belegen.
Die Genprodukte (Enzyme) der ruminalen Mikroorganismen übernehmen Aufgaben, die im Genom eines Wiederkäuers (Wirt) nicht verankert sind. Dazu gehört unter anderem der Abbau von Nahrungsbestandteilen (zum Beispiel Cellulose), die sie allein nicht zu verdauen vermögen.
Durch die Entwicklung neuer molekularbiologischer Hochdurchsatzmethoden sind die Erkenntnisse über das ruminale Mikrobiom, also die Gesamtheit der den Pansen eines Wiederkäuers besiedelnden Organismen, rasant steigend.
Die Gesamtergebnisse bestätigen, dass das ruminale mikrobielle Ökosystem direkt mit dem Pansenfermentationsmuster gekoppelt ist. Zusammenfassend kann formuliert werden:
Der vorliegende Beitrag gibt neue Einblicke in die strukturelle und funktionelle Vielfalt des Pansenmikrobioms sowie in die vielschichtig vorhandenen Interaktionen (Fütterung-Mikrobiom-Wirt).
Wir stehen allerdings erst am Anfang, die verschiedenen ruminalen Mikrobiota unserer Wiederkäuer zu erforschen.
Man könnte annehmen, dass die zugehörigen Forschungsarbeiten tiefste Grundlagenforschung – eng begrenzt auf die tierische Erzeugung – ist. Dem ist genau nicht so!
Es darf erwartet werden, dass die ruminale Mikrobiomforschung, die längst auch das Interesse der Biogas- oder Biokraftstofferzeuger geweckt hat, uns wohl noch viel Neues parat hält.
Die Koexistenz der Mikroben und ihrer wiederkäuenden Wirte – entstanden in der gemeinsamen Koevolution in Millionen von Jahren – dürfte ein Vorbild dafür sein, wie wir durch eine gezielte Nutzung von Mikroorganismen in dafür speziell errichteten Anlagen unsere Energieprobleme (mit-)lösen oder unseren steigenden Proteinbedarf zukünftig (mit-)decken könnten.
Das neue Forschungsgebiet besitzt deshalb hohe Priorität für die gesamte Lebensmittel- und Energieerzeugung auf Basis von Biomasse.
Der nachfolgende zweite Beitrag stellt die Archaeen in den Mittelpunkt der Betrachtungen.
The ruminal microbiome of cattle
Part 1: Bacteria – a central part of the ruminal microbiome
Ruminants depend on their microbiota to digest their feed.
It is therefore very tempting to prove a link between the composition and quantity of various rumen bacteria and the physiological parameters of the host.
The gene products (enzymes) of ruminal microorganisms take over tasks that are not anchored in the ruminant (host) genome such as the degradation of nutritional components (for example cellulose) that they are not capable of digesting by themselves. The development of new high-throughput methods in molecular biology is rapidly increasing the knowledge of the ruminal microbiome (the totality of microbial organisms in the rumen of a ruminant).
One important conclusion is that the ruminal microbial ecosystem is connected directly to the rumen fermentation pattern. To sum up, it can be said that:
This paper will provide new insights into the structural and functional diversity of the ruminal microbiome as well as into the multilayered existing complex interactions (feeding-microbiome-host).
However, we are now only beginning to explore the ruminal microbiota of our ruminants.
One might assume that the associated research constitutes the deepest basic research, strictly limited to livestock production. But that is just not so!
It may be expected that ruminal microbiome research, which has attracted the interest of biogas and biofuel producers for a long time, probably has many things in store for us.
The coexistence of microbes with their ruminant hosts – the result of joint co-evolution over millions of years – should be a model for how we could solve, by systematically using microorganisms in specially built facilities, our energy problems and increasing our protein needs in the future.
Therefore, this new research area has a high priority for the entire food and energy production based on biomass.
The following second report focuses its analysis on the archaea.
Le microbiome ruminal des bovins
1ère partie : les bactéries – élément constitutif central du microbiome du rumen
Pour digérer les fourrages les ruminants sont dépendants de leurs microbiotes.
Il est donc tentant de prouver qu’il existe une relation entre composition et quantité des diverses bactéries du rumen et paramètres physiologiques de l’hôte. Les produits géniques (enzymes) des microorganismes du rumen assument certaines tâches qui ne sont pas ancrées dans le génome d’un ruminant (hôte). En fait partie, entre autres, la décomposition des composants alimentaires (cellulose, par exemple) qu’ils ne peuvent pas digérer seuls.
Du fait du développement de nouvelles méthodes de biotechnologie moléculaire de pointe, les connaissances recueillies au sujet du microbiome du rumen, c’est-à-dire de la totalité des organismes colonisateurs d’un ruminant, sont en augmentation considérable.
Les résultats d’ensemble confirment que le système écologique ruminal microbien est directement couplé avec le modèle de fermentation du rumen. En résumé, on peut affirmer
Le présent article apporte de nouveaux aperçus sur la diversité structurelle et fonctionnelle du microbiome ruminal, ainsi que sur les interactions existantes (alimentation-microbiome-hôte).
A vrai dire, nous ne sommes qu’au début de la recherche concernant les différents microbiomes de nos ruminants.
On pourrait supposer que les travaux de recherche correspondants représentent la recherche fondamentale la plus profonde – étroitement limitée à la production animale. Ce n’est juste-ment pas le cas!
On est en droit de s’attendre à ce que la recherche du microbiome ruminal qui a éveillé également et depuis longtemps l’intérêt à l’égard de la production de biogaz, voire celui des producteurs de biocarburant, tient probablement à notre disposition bien des choses nouvelles. La coexistence des microbes et de leurs hôtes ruminants – apparue au cours de la coévolution commune au fil de millions d’années – devrait être un modèle qui devrait permettre de résoudre, dans le futur, nos problèmes en matière d’énergie ou nos problèmes relatifs à nos besoins croissants en protéïnes, cela par une utilisation ciblée de micro-organismes dans des installations spécialement construites à cet effet.
De ce fait, le nouveau domaine de recherche est hautement prioritaire pour ce qui est de l’ensemble de la production des produits alimentaires et celle d’énergie s’appuyant sur la biomasse.
La deuxième communication qui suit met en place les archéens au centre des considérations.
1) Als Bakteriophagen (kurz: Phagen) bezeichnet man die Virengruppe, die auf Bakterien als Wirtszellen spezialisiert sind.
2) Ein Kommensale (lat.: commensalis, "Tischgenosse") ist ein Lebewesen, das sich von den Nahrungsrückständen eines Wirtsorganismus ernährt, ohne ihn zu schädigen (Gegensatz: Parasit).
3) Nach Prof. Robert E. Hungate (1906-2004), dem Nestor der Pansen-Mikrobiologie.
4) Die ribosomale Ribonukleinsäure (kurz: rRNA) ist Bestandteil der Ribosomen. Mit den ribosomalen Proteinen zum Ribosom verknüpft, synthetisiert die rRNA von der Zelle benötigte Proteine.
5) Solche Marker sind zelluläre Makromoleküle, deren Zusammensetzung sich mit abnehmendem Verwandtschaftsgrad verschiedener Organismen immer mehr unterscheiden. Zu den wichtigsten Molekülen dieser Art zählt derzeit die 16S-Untereinheit der ribosomalen RNA. Die Basensequenz dieser RNA spiegelt evolutionäre Beziehungen unter den Organismen wieder (21, 32).
Die Größe der rRNAs wird häufig nach ihrem Sedimentationsverhalten in einer Zentrifuge (Sedimentationskoeffizient) in Svedberg (1 S = 10−13 s) angegeben.
6) In der Phylogenie ist eine operative taxonomische Einheit (OTU) eine definierbare Art (oder Artengruppen) basierend nur auf verfügbaren DNA- oder RNA-Sequenzdaten.
7) Die Domäne wurde als höchste Kategorie in der Taxonomie einführt. Die weitere Klassifikation erfolgt hier nach Stamm (Phylum), Klasse (Classis), Ordnung (Ordo), Familie (Familia), Gattung (Genus), Art (Species) und Unterart (Subspecies).
8) Bacteroidetes sind gramnegative Bakterien, die beim Abbau und Fermentieren des organischen Materials mitwirken (7, 24, 25). Da einige Arten aus der Klasse Bacteroidia in der Lage sind, pflanzliche Zellwände (Polysaccharide) zu hydrolysieren, ist es nicht verwunderlich, dass die Bacteroides besonders reichlich im Panseninhalt der Rinder zu finden sind (21, 24).
9) Die Firmicutes (lat. firmus "stark", cutis "Haut") bilden einen artenreichen Stamm. Sie gehören zu der Gruppe der grampositiven Bakterien (7, 21, 24, 25). Ein interessanter Vertreter aus diesem Stamm ist Ruminococcus flavefaciens. Ruminococcus flavefaciens gehört zur Gattung Ruminococcus in der Familie der Ruminococcaceae aus der Ordnung Clostridiales im Phylum Firmicutes (4, 6). Ruminococcus flavefaciens ist eine spezialisierte cellulolytische Bakterienarten im Pansen.
Firmicuten sind nicht nur im Pansen von Wiederkäuern zu finden, sondern beispielsweise auch Teil der humanen Darmbakterien-Community.
Die Gattung Fibrobacter (mit den beiden Arten Fibrobacter succinogenes und Fibrobacter intestinalis) aus dem bakteriellen Phylum Fibrobacteres ist gleichfalls am Abbau von Cellulose im Pansen beteiligt (7, 21, 24, 25).
10) Xylanasen sind Enzyme, die verschiedene, im Stützgerüst der pflanzlichen Zellwände vorhandene Substanzen (Xylane) aufzuschließen vermögen (6).
11) Im Allgemeinen sind Phagen sehr wirtsspezifisch.
12) Bei Kälbern mit einem schwachen Saugreflex und einem gestörten Haubenrinnenreflex (zum Beispiel nach einer Schwergeburt) kann "Pansentrinken" auftreten. Da die Tränke im unterentwickelten Pansen noch nicht richtig verdaut werden kann, sind Fehlgärungen (Gasbildung, Milchsäure) möglich, der pH-Wert fällt und eine Pansenspülung kann erforderlich werden (18, 28, 31).
13) VFA: volatile fatty acids (flüchtige Fettsäuren).
14) Die Restfutteraufnahme (RFI) wird als tierindividueller "Rest" aus der beobachteten Futteraufnahme gegenüber der (mittels gültiger Regressionsbeziehungen) vorhergesagten Futteraufnahme (aufgrund abgeleiteter Energiebedarfswerte) berechnet.
Prof. Dr. habil. Wilfried Brade
TiHo Hannover
zurzeit: Leibniz-Institut (FBN) für Nutztierbiologie Dummerstorf
Wilhelm-Stahl-Allee 2
18196 Dummerstorf
E-mail: brade@fbn-dummerstorf.de
Prof. Dr. habil. Ottmar Distl
Stiftung Tierärztliche Hochschule Hannover
Institut für Tierzucht und Vererbungsforschung
Bünteweg 17p
30559 Hannover
E-mail: ottmar.distl@tiho-hannover.de