

von PROF. DR. WILFRIED BRADE und PROF. DR. OTTMAR DISTL
Im Laufe der Evolution haben sich komplexe Symbiosen im Gastrointestinaltrakt (GIT) von Monogastriern (Wirte) und einer Vielzahl von Bakterien, Archaeen und Pilzen ausgebildet.
Da die Mehrheit der am/im Tier lebenden Mikroben im Darmtrakt zu finden ist, wird nachfolgend das Hauptaugenmerk auf das intestinale Mikrobiom gelegt.
Das Darmmikrobiom wird quantitativ von Bakterienstämmen dominiert, von denen Firmicutes und Bacteroidetes die wichtigsten sind. Intraindividuell kann die Zusammensetzung der bakteriellen Flora deutlich variieren (8).
Durch die Entwicklung neuer molekularbiologischer Hochdurchsatzmethoden sind die Erkenntnisse über das intestinale Mikrobiom rasant steigend.
Immer mehr wird deutlich, dass die mikrobielle Gemeinschaft dabei Funktionen übernimmt, die durch das Säugergenom nicht erfüllt werden können.
Nach der Geburt erfährt die Darmschleimhaut eine dramatische Veränderung mit einer ersten dauerhaften Besiedlung der Oberfläche.
(5) zeigen, dass ein (kleines) Signalmolekül (IRAK1)1 – zumindest bei Mäusen – dafür verantwortlich ist, dass sich bei neugeborenen Mäusen Mikroorganismen im Darm ansiedeln können, ohne dass die Immunabwehr des Körpers "Alarm" schlägt. So kann nach und nach ein gesundes Darmmikrobiom aufgebaut werden.
Das Forscherteam betont, dass es sich um Grundlagenforschung an Mäusen handelt, die nicht direkt auf andere Monogastrier (Mensch) übertragbar sind. Aber auch bei anderen Säugern müssen Mechanismen existieren, die eine entzündliche Abwehrreaktion auf die weitere bakterielle Besiedlung der Darmschleimhaut nach der Geburt verhindern.
Nachdem die Darmflora ausgebildet ist, tritt das Signalmolekül IRAK1 wieder in Funktion und ermöglicht so eine schützende Immunabwehr vor krankheitserregenden Keimen (5).
Im Laufe der Entwicklung des Neugeborenen adaptiert sich das Mikrobiom in seiner Zusammensetzung in Abhängigkeit von maternalen Effekten, Alter, Fütterung, therapeutischen Interventionen und weiteren Faktoren (Abbildung 1).
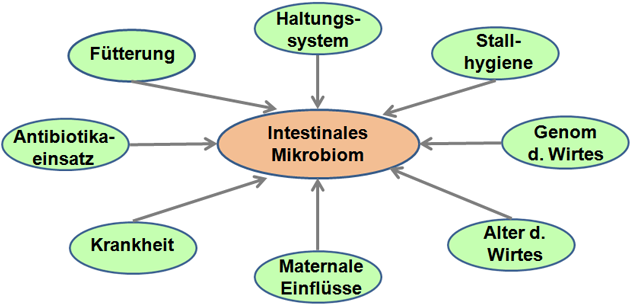
Abbildung 1: Generelle Einflussfaktoren auf das intestinale Mikrobiom.
An der Entwicklung und der mikrobiellen Zusammensetzung des Darmmikrobioms sind somit zahlreiche Faktoren gleichzeitig beteiligt, was das Erkennen detaillierter Zusammenhänge deutlich erschwert.
Die bakterielle Besiedelung des Intestinaltraktes des neugeborenen Säugers beginnt bereits während des Geburtsvorgangs mit Keimen der Vaginal- und/oder Fäkalflora des Muttertieres (10, 16).
Einige Studien sprechen sogar dafür, dass die bakterielle Besiedelung des neonatalen Magen-Darm-Trakts bereits vor der Geburt durch eine direkte Übertragung von Bakterien der Mutter auf den Fetus stattfinden kann (10, 23).
Da die erste bakterielle Besiedelung des Neugeborenen bereits im Geburtskanal oder sogar noch früher erfolgt, können bereits im Mekonium spezifische Mikroben nachgewiesen werden (1, 10, 32).
(30) belegen, dass die unmittelbare Umwelt, in der Ferkel geboren werden, ebenfalls langanhaltende Effekte auf die Zusammensetzung der Darmbakterien hatte.
Aus der Humanmedizin ist zusätzlich bekannt, dass die ersten Mikroben, die das neugeborene Baby postnatal besiedeln, vorrangig fakultative Anaerobier sind.
Die initiale Besiedelung des Darmtraktes mit vorwiegend aeroben oder fakultativ anaeroben Bakterien bereitet die Schaffung eines geeigneten Milieus für anaerobe Mikroben vor. Mit zunehmendem Alter des Säuglings nimmt die Anzahl der fakultativen Anaerobier stetig ab und die Anaerobier dominieren (1, 32).
Bei Ferkeln ist die Kolostrumaufnahme in den ersten 48 Stunden besonders wichtig, da die Plazenta des Muttertieres in der Embryonalperiode für Antikörper nicht durchlässig ist. Da antimikrobielle Peptide oder Proteine (zum Beispiel Lactoferrin) in der Muttermilch enthalten sind, inhibiert diese natürliche Milchdiät gleichzeitig das Wachstum von pathogenen Bakterien (16, 31). Sobald der heranwachsende Monogastrier neben der Muttermilch auch Breikost erhält, passt sich die Darmmikrobiota dieser veränderten Ernährung an.
Zusammenfassend kann festgehalten werden:
Das Absetzen der Ferkel im Alter von drei bis vier Wochen ist übliche Praxis. Es verursacht Stress (beispielsweise bedingt durch Umgebungswechsel, Neugruppierung, Nahrungsumstellung).
Morphologische und physiologische Veränderungen im Intestinaltrakt – insbesondere im Dünndarm (zum Beispiel Atrophie der Zotten, reduzierte Aktivität von Verdauungsenzymen und zusätzliche Störungen der Absorption) – sowie veränderte Zusammensetzungen der Darmgemeinschaften (Dysbiose) können die Folge sein (Abbildung 2).
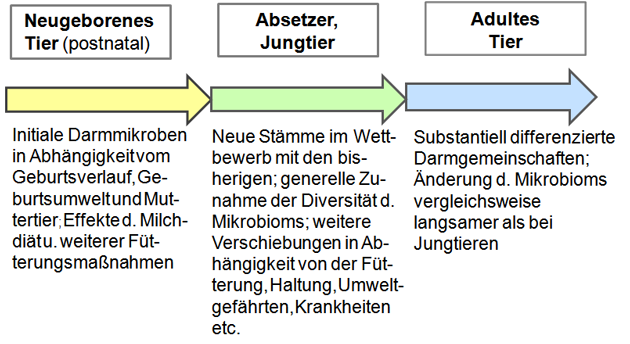
Abbildung 2: Entwicklungsstufen des intestinalen Mikrobioms beim Schwein.
Auch erweist sich die intestinale Mikrobiota adulter Tiere gegenüber externen Einflüssen, wie beispielsweise Neugruppierung oder Fütterung, im Gegensatz zu Jungtieren als generell stabiler (30, 31).
Alle Teilbereiche des Verdauungstraktes, von der Mundhöhle bis zum Rektum, sind von Mikroorganismen besiedelt.
Da der Dünn- und der Dickdarm sehr unterschiedliche physiologische Funktionen ausüben, reflektieren auch ihre jeweiligen mikrobiellen Gemeinschaften deutliche lokale Unterschiede; sowohl bei Jung- als auch Alttieren.
Zusätzlich sind innerhalb der verschieden Darmabschnitte die Bakteriengemeinschaften auf der Schleimhaut (Mukosa)2 und im Darmlumen deutlich verschieden.
Im Intestinaltrakt von Monogastriern lassen sich vier Mikrohabitate, die zum Teil auch unterschiedlich besiedelt werden, differenzieren (31: drei Habitate in den verschiedenen Mukosaschichten (die Oberfläche der epithelialen Zellen, die Mukusschicht auf den Mikrovilli und innerhalb der Lieberkühn-Krypten) und das Mikrohabitat im Lumen).
Longitudinale Analysen der mikrobiellen Gemeinschaften entlang des GIT bei Monogastriern zeigen: speziell der Dickdarm beherbergt ein enormes mikrobielles Ökosystem (Abb. 3).
Bei Monogastriern findet die mikrobielle Fermentation von Nährstoffen vor allem im Dickdarm statt. Die vorherrschenden Milieufaktoren des Dickdarmes – neutraler pH-Wert und langsamer Transport des nährstoffreichen Darminhaltes – bilden hier optimale Bedingungen für die mikrobielle Besiedlung (31).
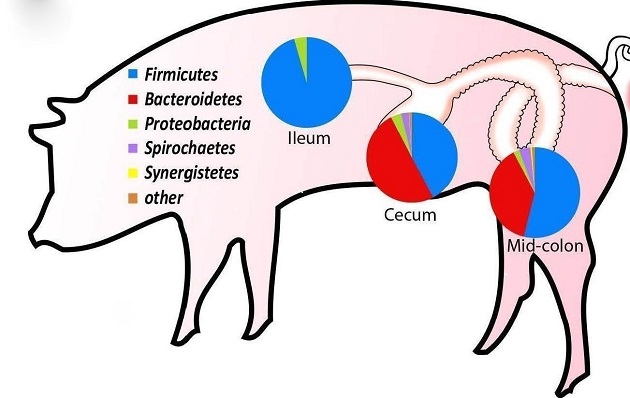
Abbildung 3: Zusammensetzung der Bakteriengemeinschaften im Darmlumen von Mastschweinen auf Phylum-Level (20).
Die Zusammensetzung der intestinalen bakteriellen Gemeinschaft beim Schwein wird vor allem durch Firmicutes, Bacteroidetes, Proteobacteria und Spirochates dominiert (Abbildung 3).
Nachzutragen bleibt, dass die Besiedlung des GIT mit Pilzen (Hefen) in gleicher Weise wie bei den Bakterien erfolgt. Die Pilze werden mit dem Futter oder Kot aus der Umgebung oral aufgenommen. Jedoch nicht alle Species passen sich den im Darm vorherrschenden Verhältnissen an. So werden einige entweder während der Magen-Darm-Passage verdaut oder finden hier keine geeigneten Bedingungen für Wachstum und Vermehrung (20, 31).
Obwohl viele Lumen-assoziierte Mikroorganismen auch auf der Schleimhaut gefunden werden, bleiben einige spezialisierte bakterielle Species auf die Mukosa beschränkt.
Die Mukosa-assoziierten Bakteriengemeinschaften enthalten beispielsweise auch Pseudomonas, Campylobacter, Bradyrhizobium, Weissella und Sutterella3 (20).
Mitglieder der Gattung Pseudomonas sind dafür bekannt, dass sie bei höherer Zelldichte Biofilme (Schleime) zu bilden vermögen, die sie wiederum gegen Fresszellen und Antibiotika schützen. Die Milchsäurebakterien Weissella spp. und die Gallesaft-abbauenden Sutterella spp. sind auch auf der menschlichen Darmschleimhaut isoliert worden; wenn auch nicht als dominante Mitglieder der bakteriellen Gemeinschaft, sondern als Teil der Milieu-spezifischen Community.
Nur drei Gattungen (Erysipelothrix, Anaerovorax und Oxalobacter) wurden von (20) mit signifikant höherer Dichte im Lumen vergleichsweise gegenüber der Besiedlung auf der Schleimhaut gefunden.
Auch die neuere Studie von (21) bestätigt das Vorhandensein bemerkenswert diverser Schleimhaut-assoziierter bakterieller Gemeinschaften in den verschiedenen Darmabschnitten mit deutlicher Abhängigkeit vom verabreichten Futter (Ca-P-Diät).
Die intestinale Mikrobiota kann wie folgt zusammenfassend charakterisiert werden:
Die vielfältigen Funktionen des Mikrobioms können in Bereiche gruppiert werden (Abbildung 4):
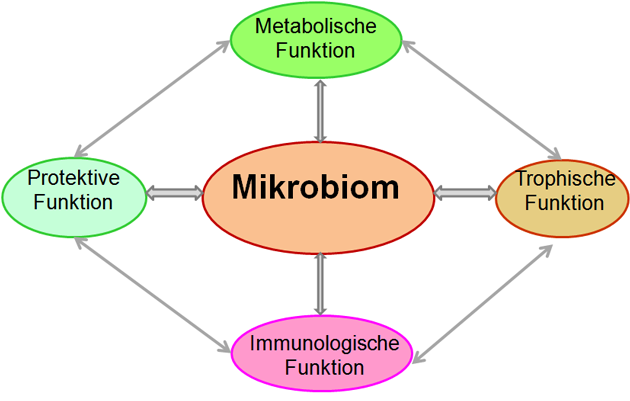
Abbildung 4: Funktionen des Mikrobioms.
Quelle: Eigene Darstellung.
Anzumerken bleibt, dass eine klare Differenzierung der genannten Funktionen nicht immer leicht möglich ist. So sind beispielsweise die protektiven und immunologischen Funktionen des intestinalen Mikrobioms eng gekoppelt (Abbildung 5).
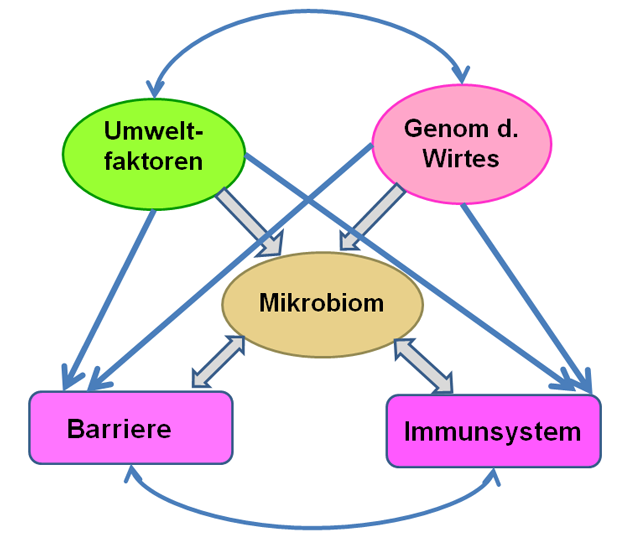
Abbildung 5: In einem genetisch suszeptiblen Tier kann das Mikrobiom sowohl zu Störungen der Barriere als auch des Immunsystems führen.
Quelle: Eigene Darstellung in Anlehnung an (4).
Darmbakterien sind vergleichsweise zu den porcinen Darmzellen in der Lage schwer verdauliche Nahrungsbestandteile, wie zum Beispiel pflanzliche Polysaccharide, zu metabolisieren. Spezialisierte Darmbakterien bilden dazu differenzierte Verdauungsenzyme (29).
Eine zentrale Funktion der intestinalen Mikrobiota ist somit der Abbau (schwer) verdaulicher Nahrungsbestandteile oder des endogenen Mukus, der vom Epithel produziert wird, einschließlich deren Metabolisierung (Abbildung 6).
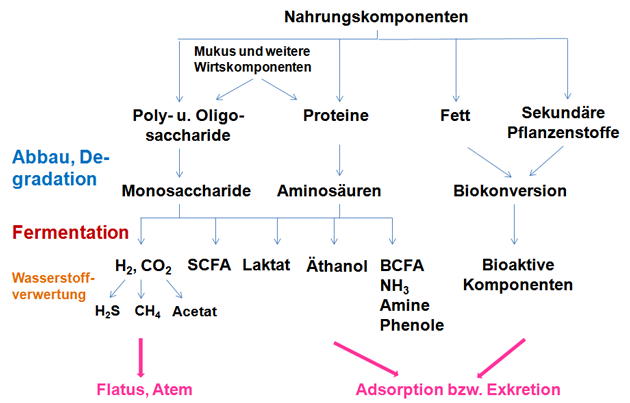
Abbildung 6: Schematische Darstellung der Degradation und Fermentation von Kohlenhydraten und Proteinen sowie der Biokonversion weiterer Nahrungskomponenten durch die intestinale Mikrobiota.
Quelle: (29) – eigene Darstellung.
Anmerkungen: SCFA = kurzkettige Fettsäuren; BCFA = verzweigte Fettsäuren.
Wesentliche Produkte der Fermentation sind Gase (H2, CO2), kurzkettige Fettsäuren (SCFA), verzweigte Fettsäuren (BCFA), Laktat, Äthanol oder auch Ammoniak (Abbildung 6). Außerdem ist die Mikrobiota in die weitere Metabolisierung der primären Gallensäure und deren Rückresorption zur Aufrechterhaltung des enterohepatischen Kreislaufs der Gallensäure sowie für die Bereitstellung von Energiesubstraten für die Kolonozysten (SCFA wie Butyrat), Leberzellen (Probionat und Laktat) oder peripheren Zellen integriert. SCFA stimuliert zusätzlich die intestinale Aufnahme von Elektrolyten (Ca, Mg) sowie Spurenelementen (Eisen) (13, 27, 29, 31).
Interessanterweise konnten Enzyme, die für den Polysaccharid-Metabolismus benötigt werden, bereits vor dem Einführen von fester Nahrung im Alter von sechs Lebensmonaten, bei menschlichen Säuglingen nachgewiesen werden (7, 32).
Neben dem Abbau der verabreichten Futtermittel oder des endogen produzierten Schleims der Epithelschicht, spielen die intestinalen Mikroorganismen auch bei der Vitaminsynthese eine wichtige Rolle für den Wirt.
Unter Trophik versteht man den Ernährungs- oder auch Wachstumszustand eines Gewebes oder Organs.
An dieser Stelle ist von Interesse, dass das intestinale Mikrobiom sowohl die Morphologie als auch die Physiologie des Darmtraktes beeinflusst (6, 11).
Durch die Kolonisation der Mikroben kommt es zu einer Verdickung der Mukosa, einer Vertiefung der Krypten und zu einer Verbreiterung und Verkürzung der Villi im Dünndarm (20, 31, 32). (6) zeigen an Ferkeln, dass Darmmikroben die Expression von Genen induzieren sowie Einfluss auf die Barrierefunktion des Epithels, die Biosynthese von Mukus oder die Etablierung des Immunsystems nehmen.
Gleichzeitig tragen die SCFAs wesentlich zur physiologischen Funktion des gesamten Intestinaltraktes bei. Die SCFAs stimulieren die Epithelzellproliferation und deren Differenzierung sowohl im Dünn- als auch im Dickdarm (29, 32).
Das intestinale Mikrobiom ist nicht nur an der Regulation und Entwicklung des GIT beteiligt, sondern auch an protektiven Funktionen oder bei der Aktivierung des Immunsystems des Wirtes (20, 32).
Kommensale Bakterien4 verteidigen den Gastrointestinaltrakt vor neuen Mikroorganismen (zelluläre Barriere). Sie verhindern zusätzlich durch Bildung von zum Beispiel antimikrobiell wirksamen Substanzen (zum Beispiel Bacteriozinen) ein Wachstum von potentiell pathogenen Keimen (Friedl, 2015).
Auch konnte wiederholt gezeigt werden, dass keimfrei aufgezogene Mäuse später sehr anfällig auf Infektionen waren (3, 13).
Es wurden zahlreiche Bakterienstämme gefunden, die die Körperabwehr stärken.
Zusammenfassend bleibt zu vermerken:
Die Mukosa stellt die Barriere zwischen innerem Milieu und intestinalem Lumen dar.
Diese Barrierefunktion hängt dabei sowohl von der Integrität der Mukosa, der mukosalen Durchblutung als auch von immunkompetenten Zellen ab.
An der gastrointestinalen Barriere sind somit verschiedenen Ebenen gleichzeitig beteiligt (Tabelle 1).
Die Epithelzellen bilden eine spezielle Schicht bestehend aus verschiedenen Zelltypen. Die Intaktheit des Epithels wird durch die interzellulären Kontakte wie den Tight Junctions und Desmosomen gewährleistet. Als biochemische Barriere wirken digestive und pankreatobiliäre Flüssigkeiten sowie eine Reihe weiterer Proteine.
Alle diese Schutzmechanismen des intestinalen Gewebes werden überwiegend der angeborenen Immunität zugeordnet und dienen letztlich der Kontrolle der mikrobiellen Kolonisation und der Prävention der transepithelialen Einwanderung von Pathogenen (19, 25).
| Tabelle 1: Bestandteile der gastrointestinalen Barriere* | ||
|---|---|---|
| Ebene | Prinzip | Beispiele |
| physikalische Barriere | intaktes Epithel | Tight Junctions, Gap Junctions |
| Darmmortalität | ||
biochemische Barriere | digestive und pankreatobiliäre Sekretion | Magensäure, Gallensäure |
| sezernierte antimikrobielle Proteine | Laktoferrin, Lysozym, Defensine | |
| weitere sezernierte Proteine | Muzine, Immunglobulin (Ig) A | |
| zelluläre Barriere | gut-associated lymphoid tissue | |
| physiologische intestinale Mikrobiota | ||
Anmerkungen: * zusammengestellt nach (25; verändert).
Das intakte Darmepithel stellt somit gemeinsam mit dem authochtonen Mukosa-assoziierten Mikrobiom eine Barriere gegen das Eindringen von pathogenen Mikroben, Antigenen und anderen Substanzen aus dem Darmlumen dar.
Neben diesen Barrieremechanismen kommt dem darmassoziierten lymphatischen Gewebe (GALT; gut-associated lymphoid tissue) eine besondere Schutzfunktion gegenüber Infektionserregern zu (Abbildung 7). Es stellt das größte lymphatische Organ des Wirtes dar (10).
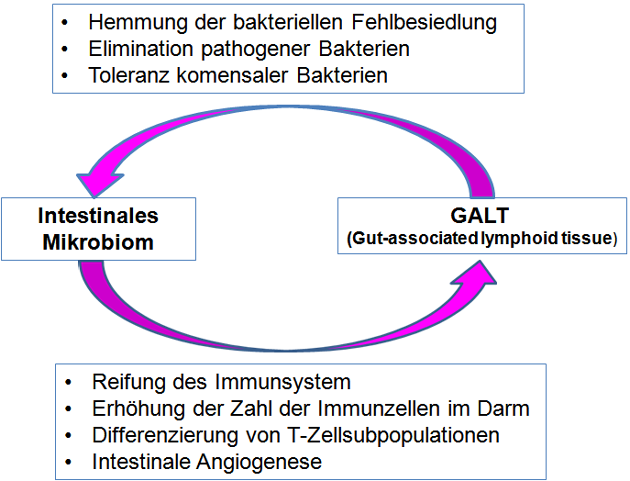
Abbildung 7: Interaktionen zwischen Mikrobiom und GALT (nach (10) – modifiziert).
Die ständige Auseinandersetzung des körpereigenen Immunsystems mit eindringenden Mikroorganismen stärkt das Immunsystem. Es darf aber auch nicht zu "Überreaktionen" führen.
Die Zusammensetzung der intestinalen Mikroorganismen unterliegt einer deutlichen Abhängigkeit von der verabreichten Nahrung, der Haltungsumwelt oder der Tiergesundheit. Unklar war bisher, ob auch das Genom des Wirtes selbst zusätzlich das Darmmikrobiom beeinflusst.
(34) untersuchten mehr als 1000 Stuhlproben5, die auch mono- und dizygote Zwillingspaare (insgesamt 416 Zwillingspaare) umfassten. Die phänotypisierten Mikrobiota monozygoter (eineiiger) Zwillinge waren sich ähnlicher als die Mikrobiota dizygoter (zweieiiger) Zwillinge. Ihre Ergebnisse zeigen somit, dass die Häufigkeit vieler mikrobieller Taxa zusätzlich von der genetischen Ausstattung des Wirtes selbst beeinflusst wird; interessanterweise aber nicht alle Mikrobenspecies in gleicher Weise.
Das Taxon mit der deutlichsten Abhängigkeit vom Genom des Wirtes war die (noch) wenig bekannte Bakterienfamilie Christensenellaceae. Sie trat gemeinsam mit anderen wirtsabhängigen Mikroorganismen einschließlich mit methanbildenden Archaeen ("Netzwerkbildung") auf. Im Gegensatz dazu waren unter anderem die bekannten und im Darm sehr häufig zu findenden Bacteroidetes nicht in gleicher Weise vom Wirtsgenom abhängig, sondern klar von der Umwelt (Ernährung) determiniert.
Zusätzliche Transplantationsexperimente (mit Mäusen) zeigten außerdem, dass mit der Verabreichung von Bakterien der Christensenellaceae-Familie auch das Übergewicht der Nager abnahm. Sie reicherten dazu die murine Darmmikrobiota mit kultivierten Bakterien aus der Familie der Christensenellaceae an (34).
Mäuse, die einen an Christensenellaceae reichen Stuhl von schlanken Menschen erhielten, blieben schlank. Mäuse, die Stuhl von adipösen Menschen erhielten, wurden ebenfalls adipös.
Die Ergebnisse bestätigen damit auch frühere Studien, in denen eine Beteiligung der intestinalen Mikrobiota an der Entwicklung der Adipositas beim Menschen nachgewiesen wurde (2, 9, 13, 17).
Es ist seit Jahrzehnten bekannt, dass der Darm-Trakt der Monogastrier Trillionen von Mikroorganismen beherbergt, die zusammengefasst heute als intestinales Mikrobiom bezeichnet werden.
Aber erst die Forschungsergebnisse der vergangenen Jahre beweisen, in welchem Ausmaß das intestinale Mikrobiom an der normalen Entwicklung und Funktion des monogastrischen Organismus beteiligt ist. Gleichzeitig belegen immer mehr Studien wie stark es auch am Auftreten einer Reihe von speziellen Erkrankungen, die keineswegs auf den GIT beschränkt sind, involviert ist (2, 17).
Der Darm-Trakt ist ein komplexes Ökosystem in einer relativ unzugänglichen Umwelt.
Der Einfluss der Darm-Mikroben auf die Entwicklung des mukosalen Immunsystems sollte nicht überraschen, da die Darmschleimhaut die größte Oberfläche zur Interaktion zwischen Antigenen der Umwelt und residenten Immunzellen bietet (4, 9, 10).
Das mukosale Immunsystem hat wiederum zwei Hauptfunktionen (4, 9, 14, 17):
Selbst zwischen Darmmikrobiom und Leber bestehen enge Wechselwirkungen: Die Leber ist einerseits Abwehrorgan gegen darmassoziierte Antigene, die über die Portalvene eingeschwemmt werden. Andererseits haben beispielsweise die von der Leber gebildeten Gallensäuren einen wesentlichen Einfluss auf die Zusammensetzung des Mikrobioms (26).
Neben dem Blick auf die bakterielle Vielfalt, konzentrieren sich aktuelle Forschungsansätze zusätzlich auch auf die funktionelle Redundanz innerhalb der intestinalen mikrobiellen Gemeinschaften und vorhandene spezielle Nischen bakterieller Spezialisten im GIT.
Ein besseres Verständnis des Zusammenhanges zwischen dem Darmmikrobiom und der Vermeidung von Krankheiten dürften auch neue Strategien, speziell in der Jungtieraufzucht oder Mast, ermöglichen (zum Beispiel durch Einsatz von Probiotika anstelle von Antibiotika in der Fütterung von Jung- oder Masttieren).
Gut belegt ist, dass durch den Einsatz von ausgewählten Antibiotika die Artenvielfalt der intestinalen Bakteriengemeinschaft des Wirtes spezifisch verändert wird (Abbildung 8).
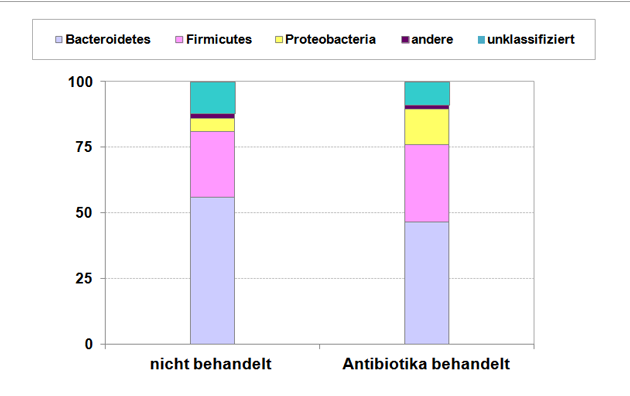
Abbildung 8: Bakterielle Zusammensetzung von Kotproben von Mastschweinen (im dritten Lebensmonat) auf Phylum-Level (ohne und mit Antibiotika-Behandlung, 14 Tage nach Behandlungsbeginn).
Quelle: Eigene Darstellung aufgrund der Ergebnissen von (20).
(20) verabreichte an drei Monate alte Mastschweine das Antibiotika ASP250 (bestehend aus: Chlortetracyclin, Sulfamethazin und Penicillin) und beobachtete die Veränderung der Mikrobiota durch Analysen von Kotproben 14 Tage nach Behandlungsbeginn. Beide Fütterungsgruppen bekamen die gleiche Standarddiät. Die unbehandelte Fütterungsgruppe jedoch ohne einen Antibiotika-Zusatz.
Die Antibiotika-Supplementierung der verabreichten Diät veränderte die bakterielle Zusammensetzung der Kotproben deutlich; mit auffallender Zunahme der Proteobacteria (Abbildung 8). Gleichzeitig wurde eine Erhöhung der Antibiotika-Resistenzgene in den behandelten Schweinen beobachtet; und interessanterweise auch einige der Resistenzgene gegenüber solchen Antibiotika, die nicht in dieser Studie verabreicht wurden. Das zeigt das Potenzial einer derartigen Fütterung auch für indirekte selektive Effekte bezüglich der Resistenz gegen Klassen von Antibiotika, die selbst nicht verabreicht wurden (20).
Einerseits schützen Darmbakterien vor Krankheiten, unterdrücken pathogene Mikroben oder bilden Vitamine. Andererseits sind sie auch Quelle für Toxine und Entzündungsmediatoren und verursachen Krankheiten (17).
Entscheidend ist, in welcher Weise der Wirt mit seinem Mikrobiom kommuniziert (2, 4, 26). Toleranz gegenüber nützlichen Bakterien und Abwehr gegen Pathogene müssen im Gleichgewicht bleiben. Dieses Gleichgewicht kann durch verschiedene Faktoren gestört sein. Zu solchen Faktoren gehören auch Medikamente; speziell Antibiotika.
Das intestinale Mikrobiom repräsentiert die Gesamtheit der im Darmkanal vorhandenen Mikroorganismen. Die vielgestaltigen Symbiosen zwischen Monogastriern (Wirte) und der intestinalen Mikrobiota oder zwischen den zahlreichen Species der mikrobiellen Darmgemeinschaft sind von wechselseitigem Vorteil.
Die Wirte liefern zum Beispiel Nährstoffe (Cellulose und andere pflanzliche Polysaccharide) und sichern optimale Lebensbedingungen für die intestinale Mikrobiota.
Die Mikroorganismen schließen für den Wirt unverdauliche Nährstoffe auf und liefern ihm darüber hinaus Aminosäuren oder lebenswichtige Vitamine.
Das intestinale Mikrobiom ist gleichzeitig an der Entwicklung des wirtspezifischen Abwehrsystems beteiligt. Auch bei der Entwicklung von speziellen (Darm-)Krankheiten – infolge einer Dysfunktion der Schleimhautbarriere (Mukosabarriere) – dürfte eine Beteiligung verschiedener mikrobieller Spezies von Bedeutung sein.
Bereits der kurzfristige Einsatz von Antibiotika verändert nachweislich die Artenvielfalt der intestinalen Bakteriengemeinschaft des Wirtes. Zusätzlich wurde eine Erhöhung der Antibiotika-Resistenzgene in den behandelten Schweinen beobachtet; interessanterweise auch bei einigen der Resistenzgene gegenüber solchen Antibiotika, die nicht verabreicht wurden.
Ein besseres Verständnis des Zusammenhanges zwischen dem intestinalen Mikrobiom und der Vermeidung von Krankheiten dürften auch neue Strategien, speziell in der Krankheitsprophylaxe, ermöglichen (zum Beispiel durch Einsatz von Probiotika anstelle von Antibiotika in der Fütterung von Jung- oder Masttieren).
Weiterer Forschungsbedarf ist deshalb dringend angezeigt.
The intestinal microbiome represents the totality of the existing gut microorganisms. The manifold symbioses between monogastric animals (hosts) and intestinal microbiota, or between the numerous species of the intestinal microbial community, are mutually beneficial.
For example, the hosts provide nutrients (cellulose, polysaccharides et cetera) and ensure the best possible living conditions for the intestinal microbiota. The microorganisms break down roughage (for the host) and also provide him with amino acids and essential vitamins.
At the same time, the intestinal microbiome is involved in the development of the host-specific immune system. Where the development of special (intestinal) diseases – due to a dysfunction of the mucosa barrier (mucosal barrier) – is concerned, participation of various microbial species might equally play a significant role.
Already short-term use of antibiotics demonstrably changes the biodiversity of the host’s intestinal bacterial community. In addition, an increase in antibiotic resistance genes can be observed in the treated pigs and, interestingly, this also holds true for some resistance genes to some antibiotics that were not administered.
A better understanding of the relationship between the intestinal microbiome and disease prevention might be provided by new strategies, specifically where the prevention of diseases is concerned, for example, through use of probiotics instead of antibiotics for the feeding of juveniles or fattening animals.
There is an urgent need for further research for this reason.
Le microbiote représente l’ensemble des organismes de l’appareil intestinal. Les symbioses multiples entre les monogastriques (hôtes) et le microbiote intestinal ou entre les nombreuses espèces de la communauté microbienne de l’intestin sont d’un intérêt réciproque.
Les hôtes fournissent par exemple des substances nutritives (cellulose et autres polysaccharides végétaux) et assurent les conditions de vie optimales pour le microbiote intestinal.
Les microorganismes dissolvent les substances non digestibles pour l’hôte et lui fournissent par ailleurs des acides aminés ou des vitamines indispensables.
Le microbiote intestinal participe également à la mise en place du système immunitaire spécifique de l’hôte. De différentes espèces microbiennes devraient également jouer un rôle dans l’évolution de maladies (intestinales) spécifiques suite à un dysfonctionnement de la barrière muqueuse.
Même une utilisation d’antibiotiques à court terme change manifestement la biodiversité de la communauté bactérienne intestinale de l’hôte. En plus, une augmentation des gènes de résistance à l’antibiotique a été observée dans les porcs en question ; il convient de noter qu’il y a même eu des gènes de résistance à des antibiotiques non-administrés.
Une meilleure compréhension des relations entre le microbiote intestinal et la prévention de maladies devrait aussi permettre de nouvelles stratégies de prophylaxie de maladies (par exemple par l’utilisation de probiotiques au lieu des antibiotiques dans l’alimentation des jeunes porcs et des animaux d’engraissement).
Il est donc urgent de mener des études plus détaillées dans ce domaine.
1) Interleukin-1-Rezeptor-Associated-Kinase1.
2) Über die Mukosa werden die durch Enzymwirkung aufgeschlossenen Nahrungsbestandteile im Dünndarm oder beispielsweise Wasser aus dem Darminhalt im Dickdarm resorbiert.
3) Sutterella in der intestinalen Mikrobiota wird in der Humanmedizin auch im Zusammenhang mit Autismus bei Kindern diskutiert (33).
4) Einige kommensale Mikroben können auch pathogenes Potential besitzen.
5) Kotproben dienen oft als ein "Proxy" (Stellvertretermaterial) für die Charakterisierung der Darmflora, weil sie leicht gesammelt werden können.
Prof. Dr. habil. Wilfried Brade
TiHo Hannover
(zurzeit: Leibniz-Institut (FBN) für Nutztierbiologie Dummerstorf)
Wilhelm-Stahl-Allee 2
18196 Dummerstorf
Email: brade@fbn-dummerstorf.de
Prof. Dr. habil. Ottmar Distl
Stiftung Tierärztliche Hochschule Hannover
Institut für Tierzucht und Vererbungsforschung
Bünteweg 17p
30559 Hannover
Email: ottmar.distl@tiho-hannover.de